前面我们介绍了各种测序技术的原理:illumina、Sanger、第三代和第四代测序技术原理,我们测序得到的是带有质量值的碱基序列fastq格式,参考基因组是fasta格式。⽤⽐对⼯具把fastq格式的序列回帖到对应的fasta格式的参考基因组序列,就可以产⽣sam格式的⽐对⽂件。把sam格式的⽂本⽂件压缩成⼆进制bam⽂件可以节省空间。如果是记录某些位点或者区域碱基的变化,就是VCF⽂件格式。如果对参考基因组上⾯的各个区段标记它们的性质,⽐如哪些区域是外显⼦,内含⼦, UTR等等,这就是gtf/gff格式。如果只是为了单纯描述某个基因组区域,就是bed格式⽂件,记录染⾊体号以及起始终⽌坐标,正负链即可。
1.fastq文件
FASTQ是基于文本的,保存生物序列(通常是核酸序列)和其测序质量信息的标准格式。其序列以及质量信息都是使用一个ASCII字符标示,最初由Sanger开发,目的是将FASTA序列与质量数据放到一起,目前已经成为高通量测序结果的事实标准。
FASTQ文件中每个序列通常有四行:
序列标识以及相关的描述信息,以‘@’开头;
第二行是序列
第三行以‘+’开头,后面是序列标示符、描述信息,或者什么也不加
第四行,是质量信息,和第二行的序列相对应,每一个序列都有一个质量评分,根据评分体系的不同,每个字符的含义表示的数字也不相同。
例如:

质量评分指的是一个碱基的错误概率的对数值。其最初在Phred拼接软件中定义与使用,对于每个碱基的质量编码标示,不同的软件采用不同的方案,目前有5种方案:
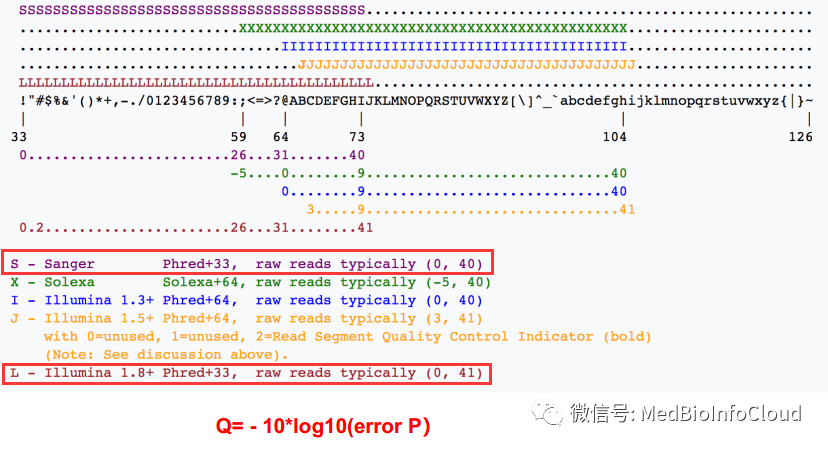
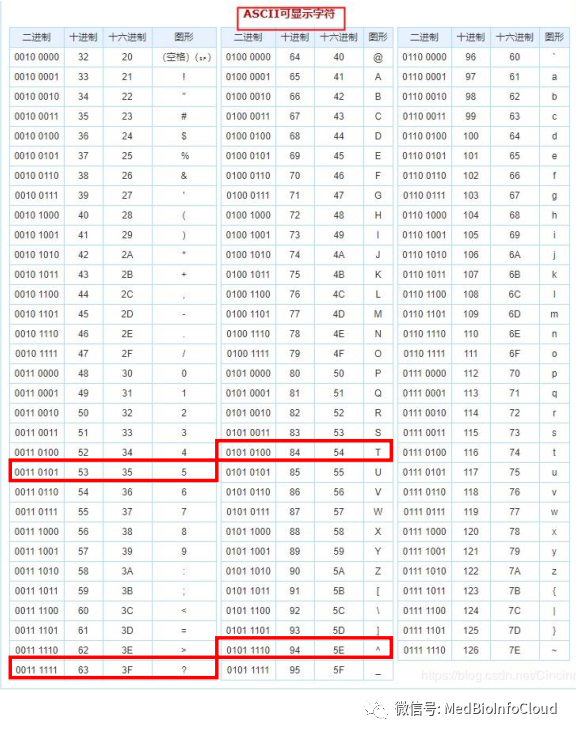
举个犊子,假设质量信息是5,这个5看成字符,不要看成数字,那么他对应的十进制数就是53,如果使用的是Sanger测序,那么质量评分采用的就是Phred+33的形式,那么Q = 53-33=20=-10lg(errorP),所以errorP = 0.01。也就计算出错误率啦,就便于我们进行质控。每一个碱基都有一个质量评分,所以第2行和第4行的位数是相同的。
2.fasta文件
FASTA格式是一种用于表示核苷酸序列或多肽序列的文本格式。其中碱基对或氨基酸用单个字母来表示,且允许在序列前添加序列名及注释。该格式已成为生物信息学领域的一项标准。
FASTA文件各行记录信息如下:
第一行是由大于号”>”开头的任意文字说明,用于序列标记,为了保证后续分析软件能够区分每条序列,单个序列的标识必须是唯一的。
从第二行开始为序列本身,只允许使用既定的核苷酸或氨基酸编码符号。通常核苷酸符号大小写均可,而氨基酸常用大写字母。注意有些程序对大小写有明确要求。一般每行60~80个字母。
核苷酸序列:

氨基酸序列:

fasta格式还是比较常见的,比如我们在NCBI查看基因的的时候通常就有fasta格式genebank格式。下面就是fasta格式的案例:
3.SAM/BAM
当我们测序得到的fastq数据map到基因组之后,会得到一个以sam或bam为扩展名的文件。这里,SAM的全称是sequence alignment/map format。而BAM就是SAM的二进制文件,也就是压缩格式的sam文件。
SAM格式文件包括头部注释部分和比对结果部分,头部分为’’可选部分’’。头部分位于比对部分之前,以“@”开头。比对部分有11列是固定的,其他多列可选。
(1)Header (标头注释部分)
@HD VN:1.0 SO:coordinate@SQ SN:chr1 LN:249250621@SQ SN:chr10 LN:135534747@SQ SN:chr11 LN:135006516...@SQ SN:chrY LN:59373566@PG ID:TopHat VN:2.0.8b CL:/home/hpages/tophat-2.0.8b.Linux_x86_64/tophat --mate-inner-dist 150 --solexa-quals --max-multihits 5 --no-discordant --no-mixed --coverage-search --microexon-search --library-type fr-unstranded --num-threads 2 --output-dir tophat2_out/ERR127306 /home/hpages/bowtie2-2.1.0/indexes/hg19 fastq/ERR127306_1.fastq fastq/ERR127306_2.fastq
(2)比对结果部分
例如这样的:
E00514:173:H3C3JCCXY:4:1124:12398:67234 337 Chr00 32904 0 150M Chr09 33498107 0 TCAATTTCACTTGAAGCTTACTTGTAGTTTCAGGCTTGGTCAAGCGCGATACAAACCATGTAGTAGGAGTCCTCCAAGTCGCCAAGCTAGGGGATCTGCTGAAAGAGGTGACAGACAAGGTAAGCAATCAGAGCTCTAAGCAATCAGTCC iieiiiii`eiiiiiiiiiiiiiiieiiiiiiiieiiiiiiiiiiiiiiiiiiiiieiiiiiiiiiiiiieiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiieiiiiiiiii`iiieeieieeieee`` AS:i:-6 XN:i:0 XM:i:1 XO:i:0 XG:i:0 NM:i:1 MD:Z:136C13 YT:Z:UU NH:i:8 CC:Z:Chr10 CP:i:18604313 HI:i:0 RG:Z:J36CK1E00514:173:H3C3JCCXY:4:1124:12398:67234 369 Chr00 32904 0 150M Chr16 2469225 0 TCAATTTCACTTGAAGCTTACTTGTAGTTTCAGGCTTGGTCAAGCGCGATACAAACCATGTAGTAGGAGTCCTCCAAGTCGCCAAGCTAGGGGATCTGCTGAAAGAGGTGACAGACAAGGTAAGCAATCAGAGCTCTAAGCAATCAGTCC iieiiiii`eiiiiiiiiiiiiiiieiiiiiiiieiiiiiiiiiiiiiiiiiiiiieiiiiiiiiiiiiieiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiieiiiiiiiii`iiieeieieeieee`` AS:i:-6 XN:i:0 XM:i:1 XO:i:0 XG:i:0 NM:i:1 MD:Z:136C13 YT:Z:UU NH:i:8 CC:Z:Chr10 CP:i:18604313 HI:i:2 RG:Z:J36CK1E00514:173:H3C3JCCXY:4:1124:12398:67234 369 Chr00 32904 0 150M Chr16 29515410 0 TCAATTTCACTTGAAGCTTACTTGTAGTTTCAGGCTTGGTCAAGCGCGATACAAACCATGTAGTAGGAGTCCTCCAAGTCGCCAAGCTAGGGGATCTGCTGAAAGAGGTGACAGACAAGGTAAGCAATCAGAGCTCTAAGCAATCAGTCC iieiiiii`eiiiiiiiiiiiiiiieiiiiiiiieiiiiiiiiiiiiiiiiiiiiieiiiiiiiiiiiiieiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiieiiiiiiiii`iiieeieieeieee`` AS:i:-6 XN:i:0 XM:i:1 XO:i:0 XG:i:0 NM:i:1 MD:Z:136C13 YT:Z:UU NH:i:8 CC:Z:Chr10 CP:i:18604313 HI:i:4 RG:Z:J36CK1E00514:173:H3C3JCCXY:4:1124:12398:67234 369 Chr00 32904 0 150M Chr17 31040767 0 TCAATTTCACTTGAAGCTTACTTGTAGTTTCAGGCTTGGTCAAGCGCGATACAAACCATGTAGTAGGAGTCCTCCAAGTCGCCAAGCTAGGGGATCTGCTGAAAGAGGTGACAGACAAGGTAAGCAATCAGAGCTCTAAGCAATCAGTCC iieiiiii`eiiiiiiiiiiiiiiieiiiiiiiieiiiiiiiiiiiiiiiiiiiiieiiiiiiiiiiiiieiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiieiiiiiiiii`iiieeieieeieee`` AS:i:-6 XN:i:0 XM:i:1 XO:i:0 XG:i:0 NM:i:1 MD:Z:136C13 YT:Z:UU NH:i:8 CC:Z:Chr10 CP:i:18604313 HI:i:6 RG:Z:J36CK1E00514:173:H3C3JCCXY:4:1212:19025:24532 409 Chr00 33538 0 150M * 0 0 GATTCCAAGTGCTGACTGATTGCTCTCTTTCTCCTTGTCTTGCAGGTAAGAACAAGGCCAAAGGAAAAGACAGGGAAAAAACATGAAATGAGATACTCTTGCTTTTAACCCTGATGATATGAGATATTCTTGCTCTAGTATAGCTTGTTT ii`e`ei[iiiiiiiiiiiiie[ieeieieiiiiieiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiiieee`` AS:i:-12 XN:i:0 XM:i:2 XO:i:0 XG:i:0 NM:i:2 MD:Z:52T33T63 YT:Z:UU NH:i:20 CC:Z:Chr01 CP:i:11331871 HI:i:0 RG:Z:J36CK1
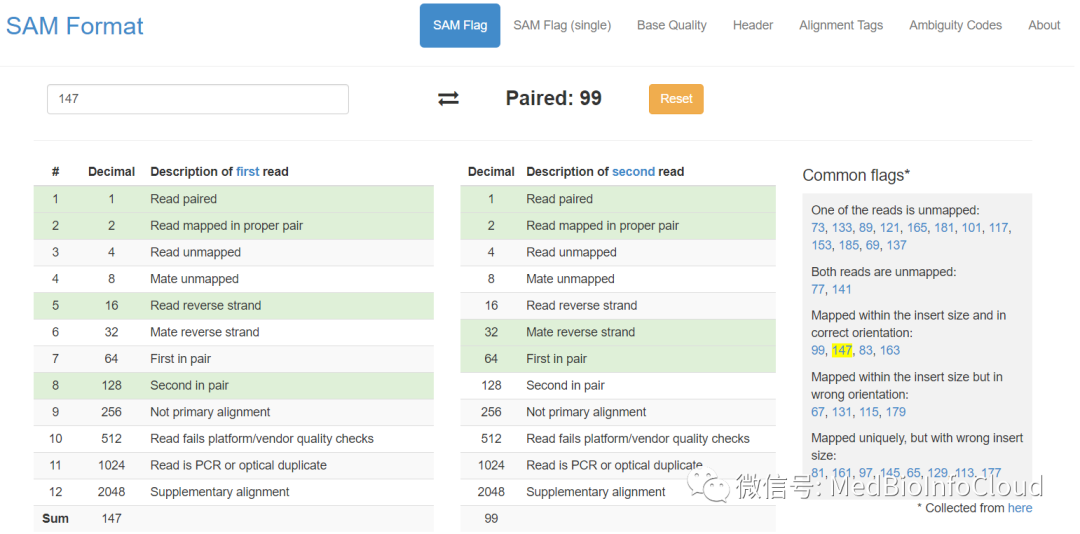
字段之间也就是列之间由Tab隔开,每一字段具体含义参考下图:

具体还可以参考官网:

由于sam格式的文件通常都非常大,所以为了节省存储空间而将sam转换为二进制格式以便于存储,也就是bam文件。sam/bam文件可以由特定的一些软件(比如samtools)来处理的,包括格式互转、排序、建立索引等操作。
4.GTF和GFF文件
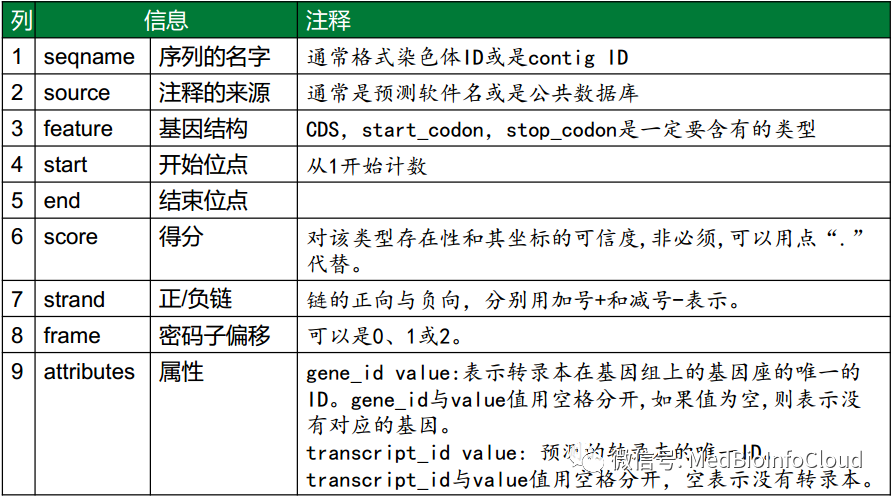
GTF全称是Gene transfer format,用于储存基因结构信息,总共有9列。关于gtf文件,我在文章:中就已经有所应用。我之前在的文章中,也是通过gtf文件进行ID转换的。
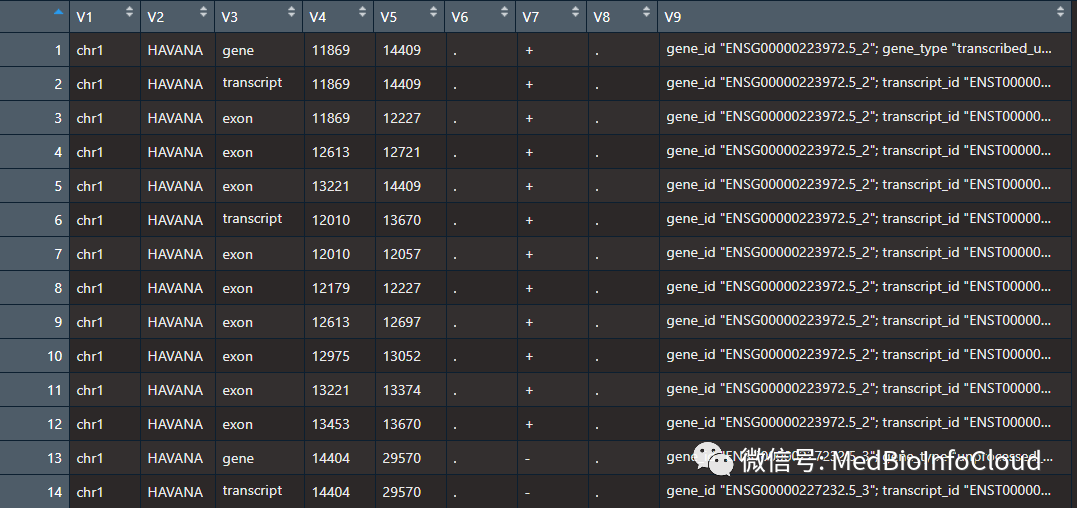
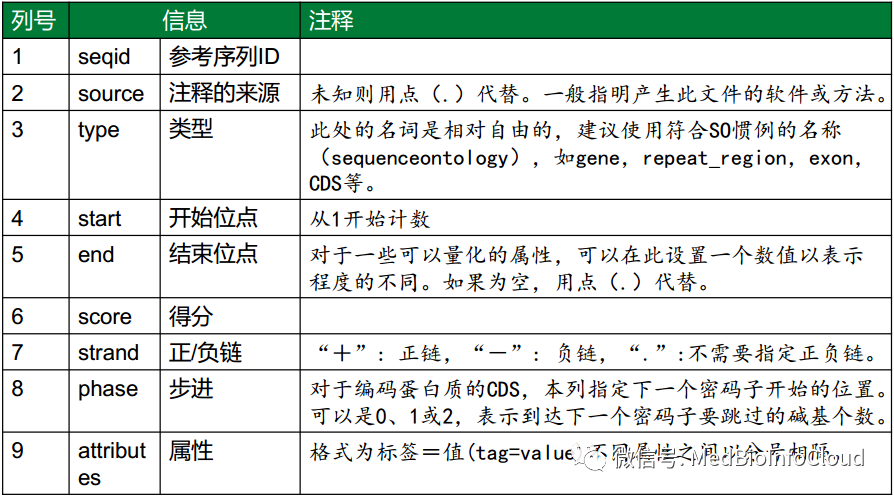
GFF全称为general feature format,这种格式主要是用来注释基因组。描述了基因组上各种特征的区间信息,包括染色体,基因,转录本等。GFF文件本质上是一个t分隔的,和gtf文件差不多,共9列的纯文本文件。
NC_000010.11 BestRefSeq%2CGnomon gene 35126830 35212958 . + . ID=gene27850;Dbxref=GeneID:1390,HGNC:HGNC:2352,MIM:123812;Name=CREM;description=cAMP responsive element modulator;gbkey=Gene;gene=CREM;gene_biotype=protein_coding;gene_synonym=CREM-2,hCREM-2,ICERNC_000010.11 BestRefSeq mRNA 35126841 35179847 . + . ID=rna82191;Parent=gene27850;Dbxref=GeneID:1390,Genbank:NM_001881.3,HGNC:HGNC:2352,MIM:123812;Name=NM_001881.3;gbkey=mRNA;gene=CREM;product=cAMP responsive element modulator%2C transcript variant 2;transcript_id=NM_001881.3NC_000010.11 BestRefSeq exon 35126841 35127193 . + . ID=id995818;Parent=rna82191;Dbxref=GeneID:1390,Genbank:NM_001881.3,HGNC:HGNC:2352,MIM:123812;gbkey=mRNA;gene=CREM;product=cAMP responsive element modulator%2C transcript variant 2;transcript_id=NM_001881.3NC_000010.11 BestRefSeq exon 35148368 35148491 . + . ID=id995819;Parent=rna82191;Dbxref=GeneID:1390,Genbank:NM_001881.3,HGNC:HGNC:2352,MIM:123812;gbkey=mRNA;gene=CREM;product=cAMP responsive element modulator%2C transcript variant 2;transcript_id=NM_001881.3NC_000010.11 BestRefSeq exon 35178889 35178986 . + . ID=id995820;Parent=rna82191;Dbxref=GeneID:1390,Genbank:NM_001881.3,HGNC:HGNC:2352,MIM:123812;gbkey=mRNA;gene=CREM;product=cAMP responsive element modulator%2C transcript variant 2;transcript_id=NM_001881.3NC_000010.11 BestRefSeq exon 35179134 35179847 . + . ID=id995821;Parent=rna82191;Dbxref=GeneID:1390,Genbank:NM_001881.3,HGNC:HGNC:2352,MIM:123812;gbkey=mRNA;gene=CREM;product=cAMP responsive element modulator%2C transcript variant 2;transcript_id=NM_001881.3NC_000010.11 BestRefSeq CDS 35148372 35148491 . + 0 ID=cds57086;Parent=rna82191;Dbxref=CCDS:CCDS7184.1,GeneID:1390,Genbank:NP_001872.3,HGNC:HGNC:2352,MIM:123812;Name=NP_001872.3;Note=isoform 2 is encoded by transcript variant 2;gbkey=CDS;gene=CREM;product=cAMP-responsive element modulator isoform 2;protein_id=NP_001872.3NC_000010.11 BestRefSeq CDS 35178889 35178986 . + 0 ID=cds57086;Parent=rna82191;Dbxref=CCDS:CCDS7184.1,GeneID:1390,Genbank:NP_001872.3,HGNC:HGNC:2352,MIM:123812;Name=NP_001872.3;Note=isoform 2 is encoded by transcript variant 2;gbkey=CDS;gene=CREM;product=cAMP-responsive element modulator isoform 2;protein_id=NP_001872.3NC_000010.11 BestRefSeq CDS 35179134 35179329 . + 1 ID=cds57086;Parent=rna82191;Dbxref=CCDS:CCDS7184.1,GeneID:1390,Genbank:NP_001872.3,HGNC:HGNC:2352,MIM:123812;Name=NP_001872.3;Note=isoform 2 is encoded by transcript variant 2;gbkey=CDS;gene=CREM;product=cAMP-responsive element modulator isoform 2;protein_id=NP_001872.3
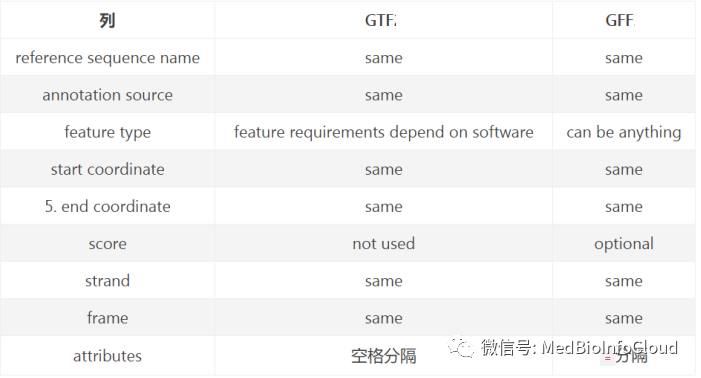
gtf与gff的比较
5.BED文件
BED文件每行至少包括chrom,chromStart,chromEnd三列必选;另外还可以添加额外的9列可选,这些列的顺序是固定的。
chr3 124792319 124792562 ENSG00000276626 RF00100 -chr1 92700819 92700934 ENSG00000201317 RNU4-59P -chr14 100951856 100951933 ENSG00000200823 SNORD114-2 +chr22 45200954 45201019 ENSG00000221598 MIR1249 -chr1 161699506 161699607 ENSG00000199595 RF00019 +
必选的三列:
例如,染色体的前100个碱基定义为chromStart = 0,chromEnd = 100,并跨越编号为0-99的碱基。
9个可选的BED字段:
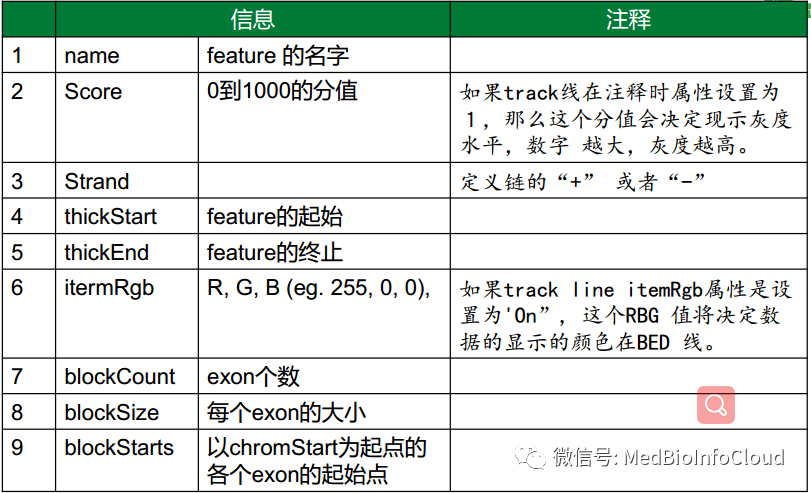
BED文件与GFF文件的区别与联系:
这里介绍一些处理bed文件的工具。

参考资料:
【1】
【2】
【3】
【4】
【5】
【6】
【7】
Ending
感谢MedBioInfoCloud公众号授权转载!

———END———
限 时 特 惠: 本站每日持续更新海量各大内部创业教程,永久会员只需98元,全站资源免费下载 点击查看详情
站 长 微 信: yjxmw518




